在蛋白与蛋白对接软件中,有两款基于快速傅里叶转换(FFT)的刚性对接软件:ZDOCK和PIPER。由于在线版的ZDOCK不会用到ZRANK重排序和聚类分析,我们就以ZDOCK为基础,结合ZRANK和聚类分析看看对挑选模型有没有帮助。
目标模型:6JBT,是PD-1与Fab的复合物结构,分辨率:2.47Å
计算过程:
从6JBT模型中分离Fab与PD1,分别保存为lig.pdb和rec.pdb。对抗原和抗体分子进行处理(去HETATM分子,补齐缺失的氨基酸,加氢等)。
用ZDOCK软件进行对接,生成2000个模型。
用ZRANK软件对对接结果进行重打分和重排序。
计算6JBT与2000个模型的Cα RMSD值、对ZDOCK和ZRANK排序结果计算Cα RMSD值并进行聚类,聚类cutoff为7.5 Å。
计算结果:
一、ZDOCK对接结果
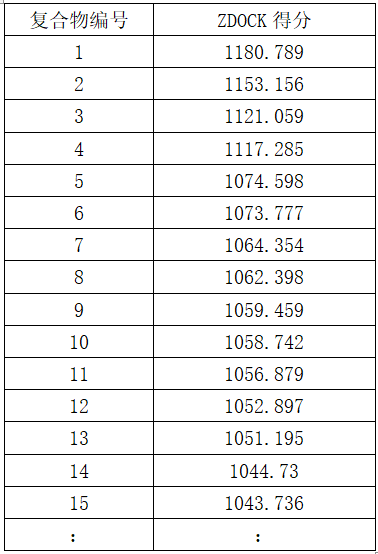
ZDOCK的输出文件已经对结果进行了排序,1为得分最高的模型,降序排列。在线版的ZDOCK一般会提供前10个对接模型。
二、ZRANK排序结果
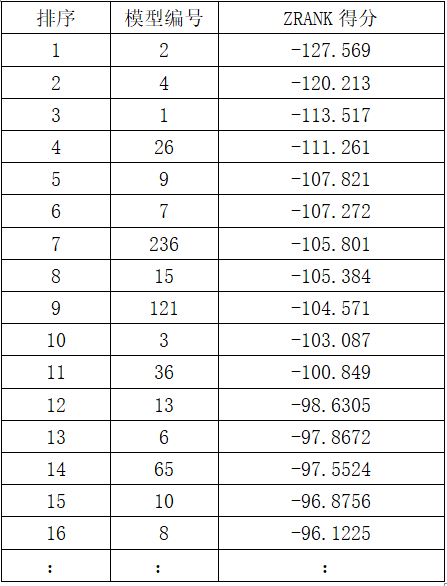
ZRANK得分越低越好,上表为升序排列。至此,ZDOCK最优的1号模型,经过ZRANK重新打分之后排在了第三位。
三、汇总分析
由于ZDOCK是一款刚性对接软件,对接结果未经过优化处理,因此我们以Cα RMSD来考察模型之间的差异。为了评价计算方法的优劣,我们采用两个参数:1. 接近天然模型的模型数量(N#)和近天然模型富集倍数(EN%)【参考文献:e8 Structure 27, 1041–1051.e1–e8, June 4, 2019】。例如,N5,N50和N500分别表示得分最高的5、50、500个模型中近天然模型的数量。
EN%=[N#/N]/[近天然模型数/总模型数]
对于刚性对接的结果,我们定义Cα RMSD在5 Å及以下的模型为近天然模型。
在2000个模型中,本次实验有79个模型与原始模型的Cα RMSD在5Å以下。
如果以ZDOCK 结果1-10号模型交付,其中近天然模型数为10个,EN%=25.31,平均RMSD为1.242Å;如果选取20个模型,那么EN%=20.25,平均RMSD=5.05 Å
如果以ZRANK结果交付前10个模型,其中近天然模型数为10个,EN%=25.31,平均RMSD为0.935Å;如果选取20个模型,那么EN%=25.31,平均RMSD=1.32 Å
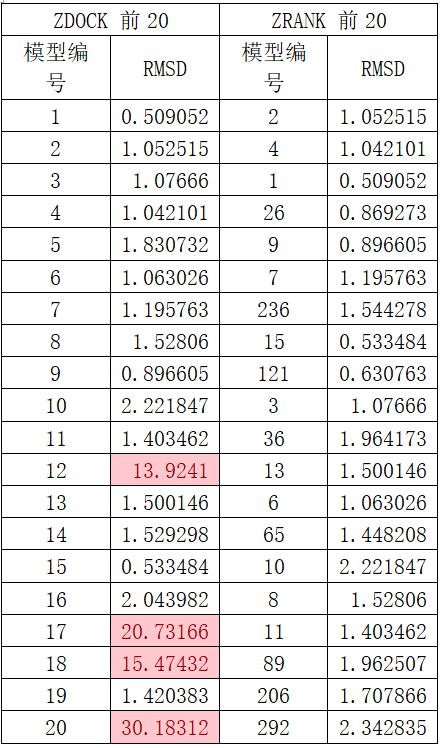
聚类结果
在选取模型的时候,有时候我们不光要考虑程序的打分情况,还要看看根据RMSD聚类的结果。最理想的情况,应该是绝大部分对接结果都集中在打分最高的模型周围。
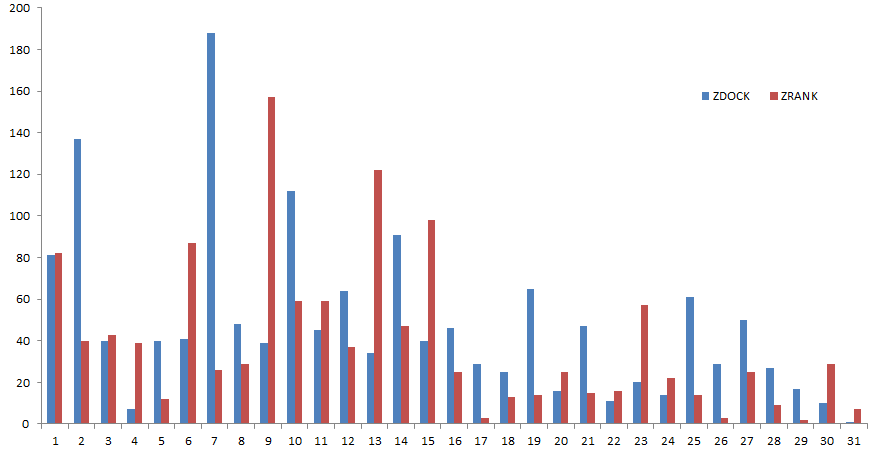
上图中,展示了前30个聚类结果。对于ZDOCK结果,第二簇的结果是否也是可以接受的,对于这一点是有疑问的(具体要看打分及结合的是否合理)。而ZRANK结果就明显更符合预期一些。
结论:在本案例中ZRANK能提高对接结果的准确性。
Souce: 纽普生物 2020-01-11