细菌导入外源基因有几种方式:
1. 转导:是指通过噬菌体将 DNA 从一种细菌转移到另一种细菌的过程。 转导可通过噬菌体的裂解循环或溶源循环进行。裂解途径是当噬菌体侵入细菌后,利用宿主菌的核糖体合成子代噬菌体,引起宿主细菌的裂解死亡,释放出子代噬菌体;溶源途径是溶源性噬菌体(温和性噬菌体)将DNA整合到宿主基因组,随细菌基因组复制而复制,并不裂解宿主菌。
2. 转化:细胞能够从周围环境中摄取DNA分子,并且不易被细胞内的限制性核酸内切酶分解时所处的一种特殊生理状态称感受态(competence)。有些细菌能够发展为自然感受态的细胞,比如枯草芽孢杆菌;有些则是非自然感受态细胞,例如铜绿假单胞菌。有些细菌经过化学试剂(如:CaCl2,RuCl等)处理后,细胞膜的通透性发生变化,成为感受态细胞,利用这种方法导入外源基因的方式称为化学转化。另外一种方式是电转化,当电流穿过细胞的时候,会在细胞膜上形成临时的孔洞,那么携带负电的质粒DNA在这个电势场中会被移动,而后恰好穿过孔洞,进入细胞。
3. 接合转移:在质粒转移过程中,供体菌与受体菌细胞通过接合作用紧密接触,质粒从供体细胞向受体转移,同时进行质粒复制,这就是接合转移的过程。接合作用由质粒或者转座子介导。
本文主要介绍下质粒介导的接合转移。
按能否自主转移,将天然存在的质粒分为两大类:
1. 自转移型质粒(接合质粒):多为低拷贝的大质粒,含有完整的转移操纵子基因(tra)和转移起始位点(oriT)。能在同种或亲缘关系近的菌体细胞间进行转移。例如:
大肠杆菌: F因子、R1、R100、R6K、RP4质粒等;F-like质粒包括:R100, R1, R6-5和ColB2-K77。 F和F-like质粒似乎仅限于密切相关的肠杆菌属,如埃希氏菌、沙门氏菌、克雷伯氏菌、志贺氏菌、肠杆菌属和柠檬酸杆菌属,宿主范围很窄。R6K的宿主范围也窄,但是近来越来越多的研究表明该质粒的衍生物可以在多种肠道和非肠道细菌中复制。RP4质粒则是一个宽宿主质粒,对它的研究非常多。
2. 可转移型质粒(mobilizable): 多为高拷贝的小质粒,如大肠杆菌的COIE1、RSF1010(8.6kb)等。由于缺少转移基因(tra),不能自主转移,但由于含有与F质粒类似的bom(或nic)位点或诱动基因mob(mobilization),因而能被带有tra基因的转移性质粒诱动而发生转移。
可转移的质粒必须包含转移原点(oriT),可以是来源于RSF1010的 mob 区(比如pBBRMCS 系列, pJN105质粒,它们都包含一个mob蛋白编码基因和mob基因的启动子,这个启动子又内含一个转移原点RSA),也可以是来源于RP4的oriT(比如pMTL8000系列质粒)等等。也就是说pBBR1MCS-5, pJN105, pMTL82151等质粒他们都是可转移质粒,但是不能自转移,它们必须需要反式元件来完成接合转移。
接合性质粒通常由四个模块组成:转移起点(oriT)区,松弛酶 (relaxase),IV型伴侣蛋白 (type IV coupling proteins, T4CP)和细菌IV型分泌系统(type IV secretion system, T4SS)。在接合转移过程中,松弛酶识别转移起始位点(oriT),结合并产生缺刻(nicking),被切割的单链进行滚环复制,T4CP识别并与ssDNA结合,随后通过T4SS从供体细胞转移到受体细胞中。
实现可转移质粒的转移可以通过双亲本杂交、三亲本杂交方式来实现。双亲本杂交需要供体细胞基因组携带RP4转移基因,如大肠杆菌菌株SM10和S17;三亲本杂交转移功能由辅助质粒提供,如pRK2013和pEVS104,它们携带RP4质粒来源的tra基因。当含有pRK2013或者pEVS104的辅助菌与含有oriT/traJ的非自转移型质粒(mobilizable plasmid, non-conjugative)的供体菌一起培养的时候,它们通过接合作用,使得供体菌也含有pRK2013或者pEVS104质粒,这样接合转移的必要基因都已齐全,使含有oriT/traJ的非自转移型质粒接合转移到受体菌中(三亲本杂交,triparental mating )。
质粒还可根据其所属的不相容组(IncQ、IncP、IncW、IncN、IncC、IncU 等)进行分类,如果两个质粒不能在宿主细胞中稳定共存,则被视为不相容(属于同一 Inc 组),这反映出它们的复制系统相同或相似。例如F质粒属于IncF、R388属于IncW、R6K属于IncX、RP4属于IncP。
目前研究的最多的接合系统有F质粒、RP4质粒和Ti质粒,它们都来源于革兰氏阴性菌。其中F质粒(致育因子)是窄宿主质粒,仅限大肠杆菌或者与大肠杆菌密切相关的菌株;RP4(抗性因子)是宽宿主质粒,可以转移到多种细菌中,包括革兰氏阳性菌;Ti质粒则存在于农杆菌中,常用于转染植物细胞。
细菌接合需要通过性菌毛来转移DNA。菌毛类型很多,根据菌毛功能可将其分为两大类:普通菌毛(Common pili)和性菌毛(Sex pili或Conjugal pili)。F-菌毛长约20微米,RP4菌毛比F菌毛要短,Ti菌毛比RP4菌毛短。
接下来我们讲讲F因子和IncP质粒RP4。
F因子又称F质粒或者致育因子(fertility factor),大小100kb左右,它可能有三种形式:F+(携带有F质粒)、F-(不携带F质粒)、Hfr(F质粒整合到细菌基因组中)。
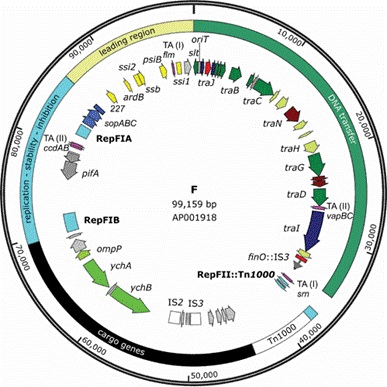
Escherichia coli K-12 F 质粒图谱[1]
F 质粒在其转移区域(tra)编码约 40 个基因。其中 15 个基因参与 F 菌毛的合成和组装。

F 质粒从供体到受体细胞接合转移过程中的生命周期示意图[2]。F 质粒骨架由编码所有参与接合转移的基因的 tra 区域(浅蓝色)、转移原点oriT(红色)、最先转移到受体细胞的前导区域(绿色)以及参与质粒复制和分割的维持区域(深蓝色)组成。(i) 接合作用的启动需要 Tra 基因的表达。产生的一些 Tra 蛋白形成 T4SS 和接合菌毛,它们将招募受体细胞并介导配对稳定。(ii) 其他 Tra 蛋白构成松弛体(TraI、TraM 和 TraY),它们与整合宿主因子(IHF)结合,通过诱导 TraI 松弛酶的裂解反应,与 oriT 结合,为质粒的转移做好准备。(iii) 松弛酶体与 IV 型偶联蛋白(T4CP)相互作用,启动 T4SS 转移 T 链。(iv,v)TraI结合的T链在受体中转移的同时,ssDNA在供体中通过滚动环复制(RCR)转化为dsDNA。(a) 进入受体后,ssDNA T 链被宿主染色体 SSB 包被,单链启动子 Frpo 采用宿主 RNA 聚合酶识别的茎环结构,启动 RNA 引物的合成。(b) TraI 对完全内化的 T 链进行环化。(c) 宿主 DNA 聚合酶识别 RNA-DNA 双链,启动互补链合成反应。(d) ssDNA 质粒转化为 dsDNA 完成后,质粒基因表达导致受体细胞表型转化为转接合细胞。
最后我们来说说IncP质粒RP4
IncP 质粒包括两个亚群 IncPK(RK2、RP1、RP4、R68 和 R18,它们是无差别的)和 IncPL(R751),由于它们能够在几乎所有革兰氏阴性菌之间转移并稳定地维持自身,因此被广泛研究。它们还能促进 DNA 从革兰氏阴性菌向革兰氏阳性菌以及酵母菌的接合转移。
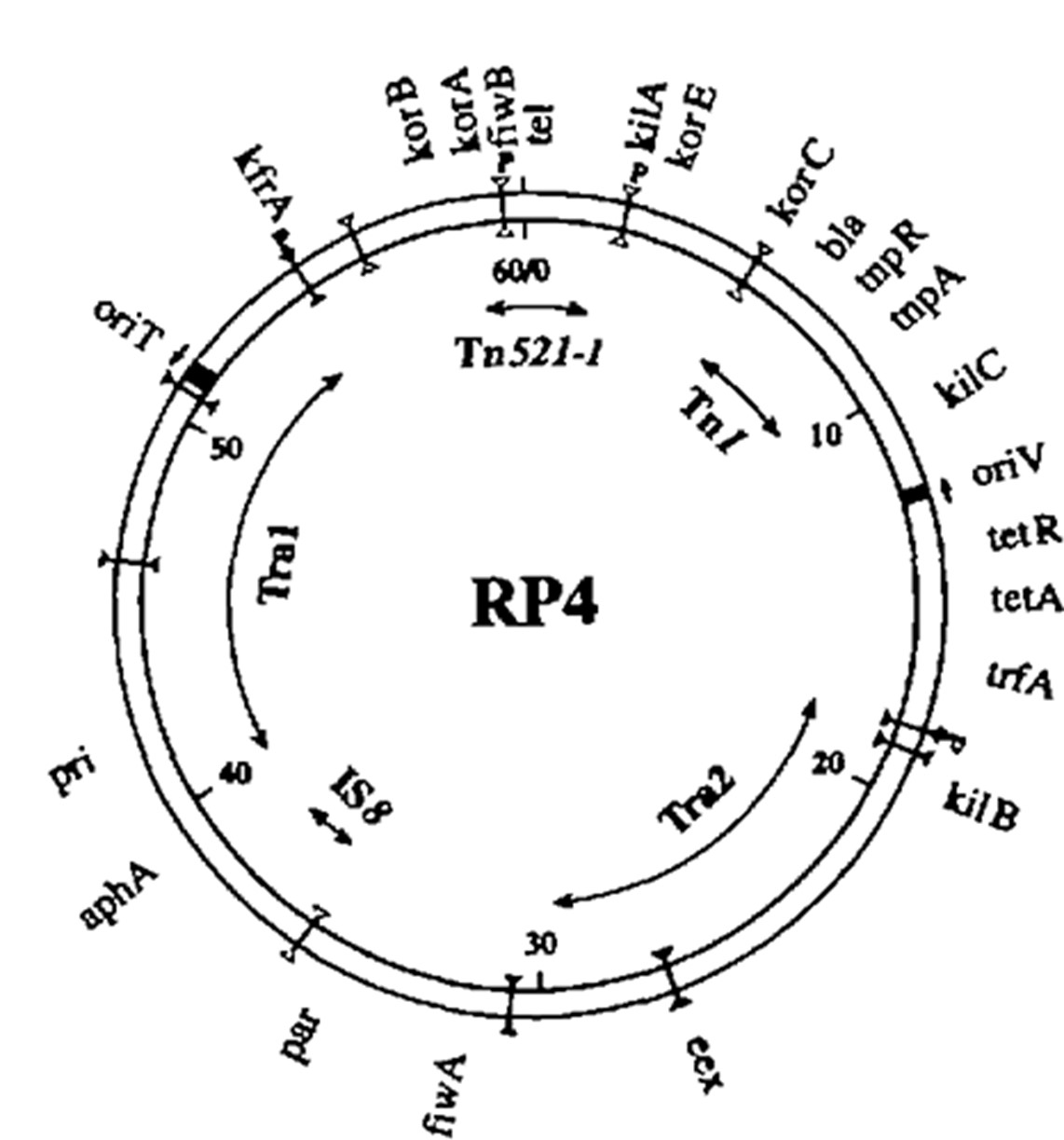
RP4序列图谱:https://www.ncbi.nlm.nih.gov/nuccore/PP591959.1
RP4质粒全长60Kb左右,转移区分为Tra1和Tra2两个操纵子。OriT在Tra1操纵子内。TraJ结合在邻近缺刻位点的oriT的反向重复序列上。
两个常用的接合辅助质粒:pRK2013和pEVS104,是IncP型辅助质粒,可以将基因转移到:不动杆菌、农杆菌、芽孢杆菌、伯克霍尔德菌、弯曲杆菌、拟甲色球藻、脱硫弧菌、嗜盐单胞菌、念珠藻、假单胞菌、根瘤菌、沙门氏菌、鞘氨醇单胞菌、链霉菌、弧菌、黄单胞杆菌等等。
pRK2013来源于RK2质粒,带ColE1复制原点;pEVS104是pRK2013的改进版,带R6Kγ复制原点。它们都是自转移型质粒。[3]
pRK2013和pEVS104骨架部分大部分相同,该骨架包含接合转移和配对形成的基因(Tra1 和 Tra2 区域;tra 和 trb 基因)。此外,还存在转录调节基因、质粒分离基因和质粒维持基因(korAB、incC、kfrABC)。质粒 RK2 的 kilA 基因座(klaABC 基因)大部分已被移除,但 klaC 基因片段仍然存在。
pEVS104 缺乏 pRK2013 中的转座子,而这种转座子能诱变受体菌株。此外,pEVS104 不能在受体细菌中复制;pRK2013 通过 ColE1 起源复制,但 pEVS104 使用 R6Kγ 复制原点,只能在含有pir基因的工程大肠杆菌供体细胞中复制。在构建 pEVS104 时,含有 ColE1 复制原点、 Tn903 转座子和编码 Colicin E1(cea)及其同源免疫基因(imm)的片段被移除。pEVS104 相对于 pRK2013 有两个插入部分:一个 R6K γ 复制原点和一个 kanR 基因相连的第二个 oriT,kanR 基因用于抗性筛选。
参考文献
1. Koraimann G.2018.Spread and Persistence of Virulence and Antibiotic Resistance Genes: A Ride on the F Plasmid Conjugation Module. 8:10.1128/ecosalplus.ESP-0003-2018.https://doi.org/10.1128/ecosalplus.esp-0003-2018
2. Virolle C, Goldlust K, Djermoun S, Bigot S, Lesterlin C. Plasmid Transfer by Conjugation in Gram-Negative Bacteria: From the Cellular to the Community Level. Genes (Basel). 2020 Oct 22;11(11):1239. doi: 10.3390/genes11111239. PMID: 33105635; PMCID: PMC7690428.
3. Hall A, Donohue T, Peters J. Complete sequences of conjugal helper plasmids pRK2013 and pEVS104. MicroPubl Biol. 2023 Jul 13;2023:10.17912/micropub.biology.000882. doi: 10.17912/micropub.biology.000882. PMID: 37521139; PMCID: PMC10375283.
Souce: 纽普生物 2024-08-05