转座子/可转座元件(TE、transposon或跳跃基因)是一种 DNA 序列,可改变其在基因组中的位置,有时可产生或逆转突变,改变细胞的遗传特性和基因组大小。转座通常会导致相同遗传物质的复制。
一般来说,按照转座方式的不同,可将转座子分为三大类:I型转座子(Class I elements),II型转座子(Class II elements)以及Helitron转座子。

图1. 三类转座子[1]
I型转座子又叫反转座子(retrotransposon)。根据反转座子的转座机制,人们形象地称其为“复制-粘贴”型转座原件。反转座子在转座时,会先以DNA为模板,在RNA聚合酶II的作用下,转录成一段mRNA,然后再以这段mRNA为模板反转录成cDNA,最后在整合酶的作用下将这段cDNA整合到基因组上新的位置。
II型转座子也叫做转座子(transposon),与反转座子“复制-粘贴”的机制不同,II型转座子转座的机制被称为“剪切-粘贴”。在转座酶的作用下,II型转座子从原来的位置解离下来,再重新整合到染色体上。而原来的位置由于转座子解离形成的断链,在DNA修复的机制下得以修整。最终的结果是,原来的位置少了一段转座子序列,而新位置多了一段转座子序列。和反转座子一样,II型转座子也可分为自主型和非自主型。非自主型转座子不具有转座必须的所有的成分,因此依赖于自主型转座子。常见的II型转座子包括:来源于细菌的Tn3、Tn5、Tn10;来源于昆虫的piggyBac(PB)、Mariner转座子;来源于鱼的睡美人转座子(Sleeping Beauty,SB)和Tol2 转座子;
Helitrons 转座子是近年来发现的一种新型 DNA 转座子,最初是利用基于重复序列的计算方法在拟南芥基因组中鉴定出来的。后来发现,大多数植物和许多动物基因组中都携带 Helitrons 转座子。Helitrons 转座子具有典型的 5'TC 以及 3'CTRR(R为A或G)末端,并在3'末端上游约 15~20bp 处有一个茎环结构,是转座子的终止信号。Helitrons 转座子转座后,通常插入 AT-rich 区域的 AT 靶位点。和反转座子和转座子不同,Helitrons 通过滚环(rolling circle)的方式进行转座。并且,在滚环复制的转座过程中经常捕获和携带基因片段,可导致基因拷贝数的变化,也会在一定程度上促进基因组的进化。
Tn5转座子
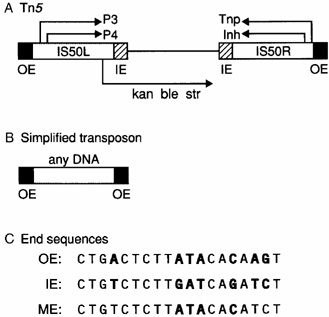
图2. Tn5转座子结构[2]
Tn5转座子由编码三个抗性基因的核心序列(kan、ble、str)和两条倒置的IS50序列组成,其中IS50R和IS50L的序列高度同源,含有编码转座酶(TnP)以及转座阻遏蛋白(lnh)的基因,但IS50L的一个碱基存在突变导致翻译提前终止,因此只有IS50R可以产生正常的有活性的TnP和lnh。IS50具有19bp的倒置末端(外末端OE和内末端IE),两倒置末端有7个bp不同(如图C中加粗碱基),此倒置末端是转座酶(Tnp)的作用位点,其中IE在只有在没有Dam-DNA甲基化的情况下才可被 Tnp 识别。ME是IE和OE的嵌合体,OE和IE均为次优序列,而ME是超活性序列。
OE: CTGACTCTTATACACAAGT
IE: CTGTCTCTTGATCAGATCT
ME: CTGTCTCTTATACACATCT
研究人员发现,整个转座子序列并不是转座必须的,只需转座子的末端核心序列(OE/IE/ME),转座酶便能将该部分序列插入并连接至基因组内(如图B)。Tn5转座酶的插入位点具有很高的随机性,因此被广泛的用于体外转基因(外源基因整合到宿主细胞)和二代测序建库等领域。Tnp结合和插入也有一定的偏好性,其首选的DNA靶序列是A-GNT(T/C)(A/T)(A/G)ANC-T,其中N是任何核苷酸。
野生型Tnp是一种非常不活跃的蛋白质,纯化的野生型Tnp在体外无转座活性,体内转座频率很低。因此为了增强Tnp的活性,实际应用的一般都是Tn5转座酶的突变体形式,比如:L372P、E54K、E110K和E345K、P242A或P242G。
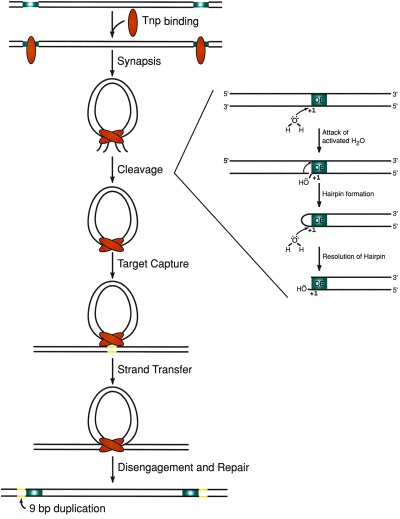
图3. Tn5转座的原理
piggyBac转座系统
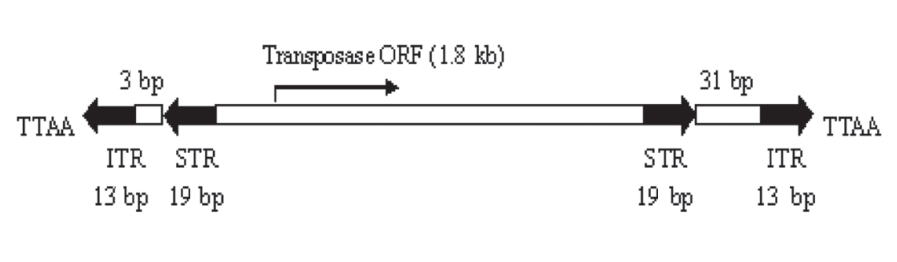
图4. PB转座系统的结构
PB全长2472 bp,两端各有一个13 bp和19 bp的反向末端重复序列(inverted terminal repeat, ITR),中间编码一个长594个氨基酸残基的转座酶。根据转座酶的转录方向来区分转座子的两个末端,两端最外侧各有长13 bp并且对称的ITR,内侧各有一段间隔区(spacer),但并不对称(左端长3 bp,右端长31bp),再靠内侧是各长19 bp且对称的亚末端反向重复序列(sub-terminal inverted repeat, STR)。反向重复序列的5'有2~3个C碱基,对应3'端的G碱基在选择剪切位点过程中均起作用。研究发现只有左侧LTR长于311 bp,并且右侧LTR长于235 bp的PB转座子才具有转座活性。转座子左右两个末端组合研究表明仅有“左+右”及“右+左”具有转座活性,前者转座活性为后者的4.6倍。
PiggyBac转座子在发现后经过了一系列优化和改造的过程,如转座酶密码子优化及突变,以及两端ITR序列的简化,最后形成了一套完整的PiggyBac载体系统。PiggyBac载体系统成员主要有:一个辅助质粒:编码转座酶;一个转座子质粒:含优化的两端亚末端反向重复序列,中间是被转座区域,可插入我们想转座到宿主基因组中的目的基因序列。
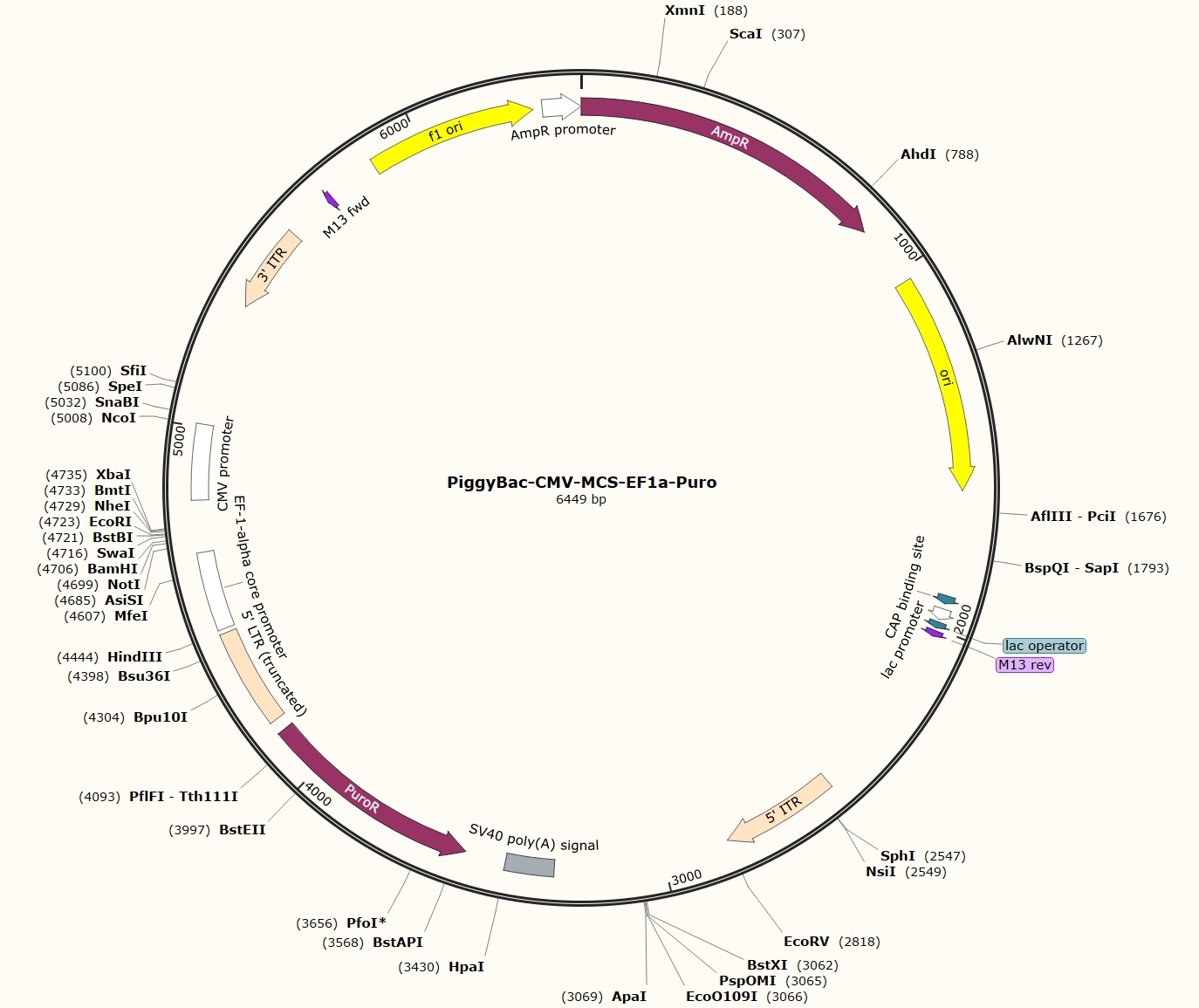
图5. 一个典型的PiggyBac转座子质粒图谱
在PiggyBac转座酶的存在下,5’ITR和3’ITR及他们之间的DNA片段会被整合到基因组中含TTAA序列的位点。
实验时需同时将辅助质粒和转座子质粒共同转化靶细胞,辅助质粒编码的转座酶识别转座子质粒两端的ITR序列并切割,释放的被转座区被转座酶整合到宿主基因组中含TTAA序列的位点,并在被转座区两端出现TTAA重复序列。piggyBac可以转座20kb以内的片段。
睡美人转座子和Tol2转座子
“睡美人”转座子是Tc1/mariner转座子超家族中的一员。Tc1/mariner转座子原本是人类、动物和鱼类等脊椎动物天然的遗传成分,但是经过亿万年的进化历程,大多数转座子失去了活性。1997年,明尼苏达州大学的Ivics等收集了来自8个不同鱼类品种中的12个失活的鲑鱼科亚家族Tc1类转座酶基因的序列,经过多重序列比对,并进行了分子重建,使其恢复跳跃能力。这项研究使沉睡了一千多万年的转座子再次被激活,因此被命名为“睡美人”。
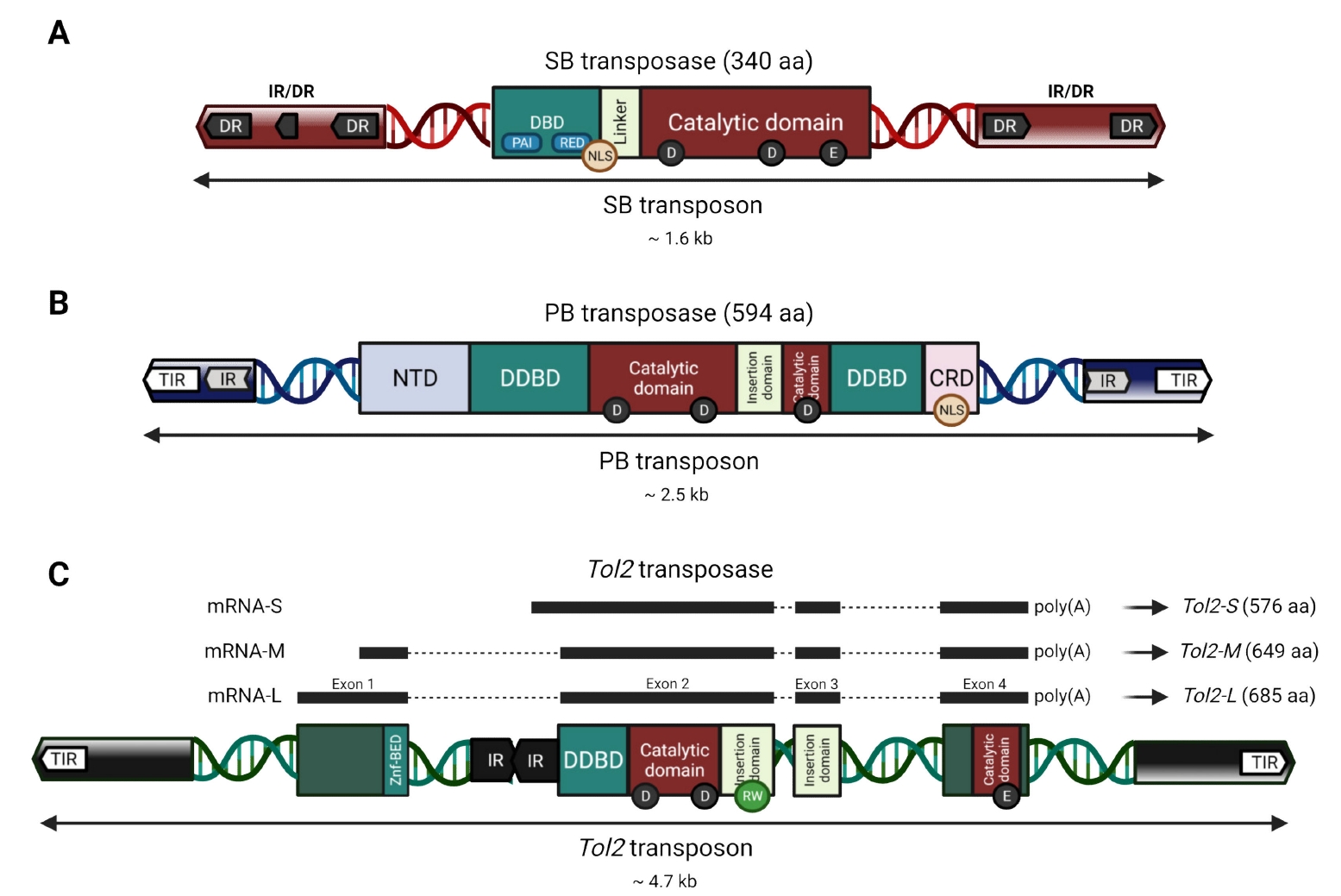
图6. SB、PB和Tol2转座子[3]
SB转座子由两部分组成:转座酶基因和能够被转座酶基因所识别的两端反向重复序列,结构如图6A所示。转座酶基因的开放式阅读框编码340个氨基酸的蛋白质, 它可以与两端的反向重复序列相结合,促进转座的发生。两端的反向重复序列长约为230 bp, 由外侧的32 bp反向重复序列(IR)内侧与IR相似的同向重复序列(DR)及两者间相距165 bp-166 bp的片断等3个部分组成。DR是转座酶的结合区域,是转座子的严格保守区。一般情况下,SB转座子能特异性地整合到基因组中的TA双核苷酸位点。
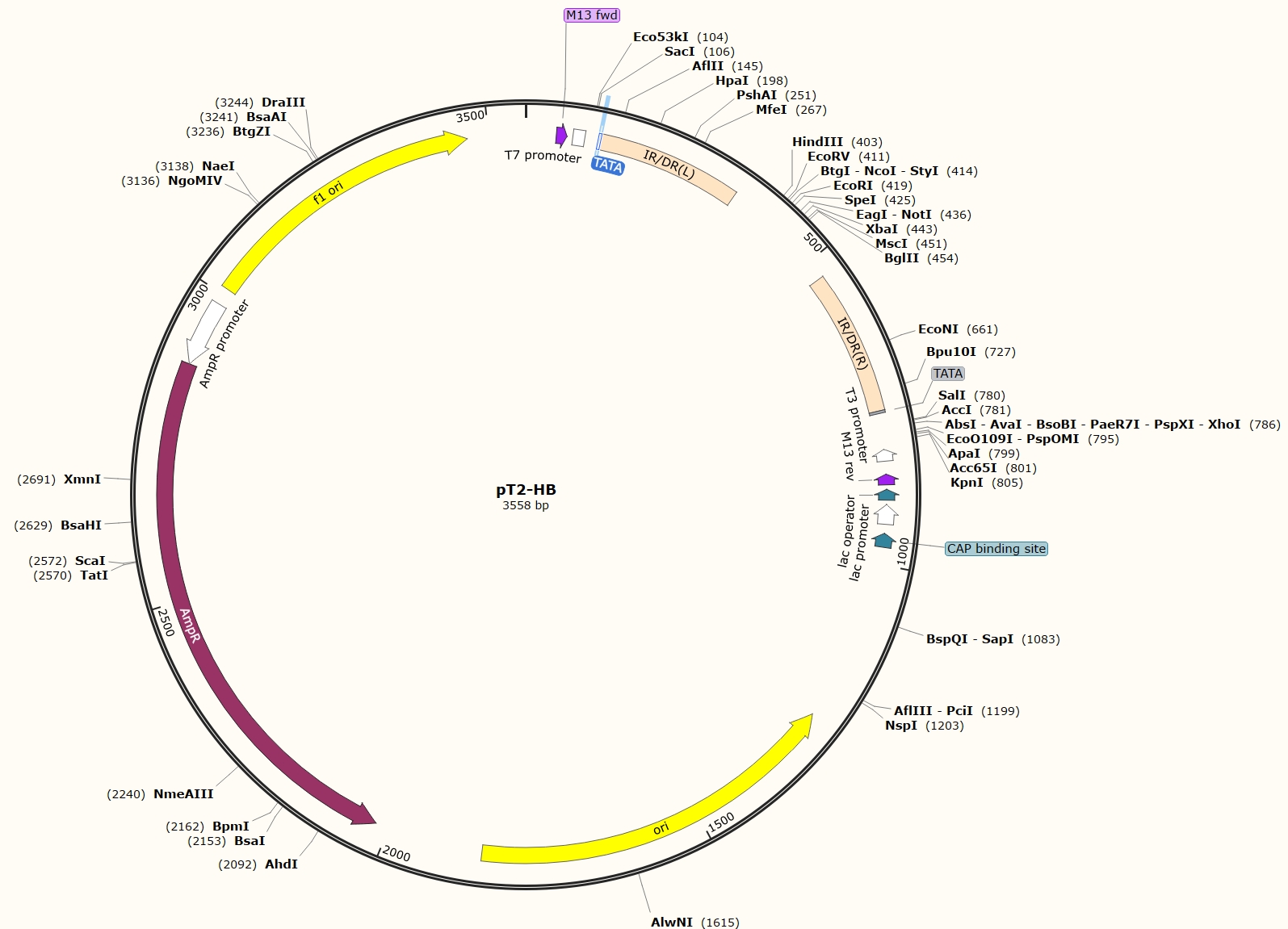
图7. 一个典型的SB转座子质粒图谱
在SB转座酶的存在下,IR/DR(L)和IR/DR(R)及他们之间的DNA片段会被整合到基因组中含TA序列的位点。
Tol2 是在青鳉鱼基因组中发现的一种具有自主性的转座子元件。它编码转座酶,催化 Tol2 转座子结构中 5'端 200 bp 和 3'端 150 bp 序列发生转座反应。Tol2转座子结构如图6C
Tol2转座子具有不定向性,即在基因组中是随机插入,并无序列特异性。
Tol2系统包含两个载体:负责编码转座酶的质粒和转座子质粒[包含两个反向末端重复序列(ITRs)以及两者之间的转座区域。
转座酶识别转座子的两个ITR并将被转座区和两个ITR元件插入到宿主基因组中。
Tol2通过“剪切-粘贴”机制进行基因整合,在每一个插入位点都会产生一个8 bp的重复序列。
| Sleeping Beauty (SB) | piggyBac (PB) | Tol2 | |
| 物种起源 | 鲑鱼科鱼类 | 甘蓝蠖度尺蛾 | 青鳉鱼 |
| 分类 | Tc1/mariner超家族 | PB超家族 | hAT超家族 |
| 可转座元件 | -1.6kb长 | -2.5kb长 | -4.7kb长 |
| 末端区域 | 约230bp的IR/DRs | 35-63bp,带有外部TIRs和内部亚末端IRs | 150-200bp,包含TIRs和亚末端区域 |
| 转座酶 | 340aa | 594aa | 649aa(最活跃异构体) |
| 足迹 | CAG | 无 | 可变 |
| 靶位点偏好 | TA | TTAA | 弱共识序列TNA(C/G)TTATAA(G/C)TNA |
| 靶位点重复 | TA | TTAA | 8bp |
| 在物种中的活性 | 各种脊椎动物 | 脊椎动物、昆虫、植物、酵母 | 各种脊椎动物 |
| 在人类细胞中的效率 | 与逆转录病毒载体相当 | 与逆转录病毒载体相当 | 低于PB和SB |
| 装载容量 | >100kb | >100kb | >100kb |
| 过度生产抑制 | 是 | 在一定程度上 | 低于PB和SB |
| 整合谱 | 接近随机 | 偏向于TSSs、CpG岛和DNasel超敏位点 | 偏向于TSSs、CpG岛和DNasel超敏位点 |
| 最常见的载体骨架 | pT2 | pXL-Bacll | pTol2, miniTo/2 |
| 最活跃的转座酶 | hySB100X | hyPB | hTol2-M |
| 转座子递送载体 | 质粒DNA、pFAR、MC、非整合型病毒载体、纳米颗粒 | 质粒DNA、dbDNA、非整合型病毒载体、纳米颗粒 | 质粒DNA |
| 转座酶递送载体 | 质粒DNA、mRNA、SNIM RNA、重组蛋白(hsSB)、非整合型病毒载体、纳米颗粒 | 质粒DNA、mRNA、非整合型病毒载体、纳米颗粒 | 质粒DNA、mRNA、重组蛋白(His-Tol2) |
| 临床试验 | 是 | 是 | 否 |
参考文献
Lisch D. How important are transposons for plant evolution? Nat Rev Genet. 2013 Jan;14(1):49-61. doi: 10.1038/nrg3374. PMID: 23247435.
Reznikoff WS. Tn5 as a model for understanding DNA transposition. Mol Microbiol. 2003 Mar;47(5):1199-206. doi: 10.1046/j.1365-2958.2003.03382.x. PMID: 12603728.
Sandoval-Villegas N, Nurieva W, Amberger M, Ivics Z. Contemporary Transposon Tools: A Review and Guide through Mechanisms and Applications of Sleeping Beauty, piggyBac and Tol2 for Genome Engineering. Int J Mol Sci. 2021 May 11;22(10):5084. doi: 10.3390/ijms22105084. PMID: 34064900; PMCID: PMC8151067.
Souce: 纽普生物 2024-08-09