传统的基因组整合技术,如逆转录病毒、转座子等载体系统存在诸多问题,不利于基因功能的分析。一方面,其整合序列特异性低,整合位点无法预测;另一方面,可能还存在插入突变等情况,进而影响基因表达。
为了提高整合的特异性,减少整合位点的数量并使整合位点的位置更具可预测性,下面将介绍一种来源于链霉菌噬菌体的整合酶系统 ——ΦC31 整合酶系统。
位点特异性重组酶分为酪氨酸和丝氨酸重组酶两个家族,酪氨酸重组酶家族主要存在于原核生物中,成员包括 λ 噬菌体整合酶、Cre 重组酶、Flp 重组酶等。
ΦC31整合酶是一种源自链霉菌噬菌体的位点特异性重组酶,属于丝氨酸催化的重组酶家族。ΦC31整合酶系统主要由整合酶和其对应的attB(细菌附着点)、attP (噬菌体附着点)位点组成。
在自然界中,ΦC31整合酶催化链霉菌基因组中的attB位点和ΦC31噬菌体基因组中的attP位点之间进行精确的重组。当整合酶与 attB 和 attP 位点结合后,进而通过催化attP和attB位点的重组将噬菌体核酸整合到宿主染色体中。在整合过程中,ΦC31整合酶与attP和attB结合形成两个二聚体,引导两个位点之间结合形成四聚体,催化丝氨酸残基进攻DNA链,催化链置换形成新的 attL 和 attR 位点。这个过程将外源基因稳定地整合到宿主基因组中,实现了基因的转移和表达。与转座子介导的基因整合不同的是,ΦC31整合酶催化的整合是单向不可逆的。
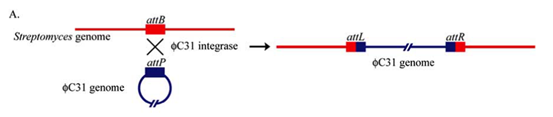
Fig1. 自然界ΦC31 整合酶介导的重组示意图
ΦC31 系统已开始用于植物(如烟草、小麦和拟南芥)、 无脊椎动物(如果蝇、蚊子和蚕)、脊椎动物(鱼和两栖动物、鸟类、哺乳动物)。
假attP 位点
在将 ΦC31 整合酶应用于真核细胞时,发现其在自然状态下,通过与噬菌体基因组中的 attP 位点和宿主链霉菌基因组中的 attB 位点相互作用,催化噬菌体基因组整合到宿主细菌基因组中。由于 attP 和 attB 位点的最小尺寸分别为 39 和 34 个碱基对,推测与病毒和转座子整合位点的频率相比,真核生物基因组中可能以相对较低的频率存在与野生型 ΦC31 attP 和 attB 位点功能相似的简并序列,即假attP 位点和假attB 位点。
通过对大量整合事件的序列分析,发现人类基因组中存在 “假” attP 位点,这些位点有一个高度显著的共识序列,与 attB 位点的同一性为 50%,与野生型 attP 的同一性为 64%。
估计人类基因组中总共有约 370 个整合位点,不同细胞系中的整合位点基本一致,但在不同组织来源的细胞中存在一些差异。
实验发现当质粒携带 attB 位点时,φC31 整合酶能将其高效整合到哺乳动物基因组的假attP 位点,而携带 attP 质粒的整合效率较低,可能是由于 attB 序列中含有 CpG 甲基化位点。
整合位点大多在基因间区域,少数在内含子和外显子区域,且整合酶倾向于整合到转录活跃的区域,这使得基因表达通常较强且持久。基因组中的整合事件大多略有不精确,伴随小的缺失或插入,但约 10% 的整合事件存在较大偏差,包括染色体缺失和易位等,但这些事件也由 φC31 整合酶介导,且大多数发生在可识别的假位点。
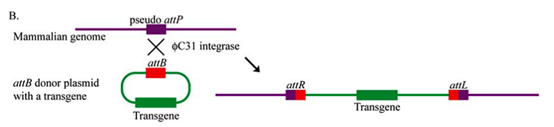
Fig2. 哺乳动物细胞ΦC31整合酶介导的重组示意图
链霉菌attB序列:GTGCCAGGGCGTGCCCTTGGGCTCCCCGGGCGCG
噬菌体attP序列:CCCCAACTGGGGTAACCTTTGAGTTCTCTCAGTTGGGGG
实验过程中,需要同时将编码ΦC31 整合酶的质粒和目的基因的质粒转染至到宿主细胞中。
pSET152载体(带有attP)能够将外源基因整合进入链霉菌基因组中,是链霉菌基因组高表达特定基因时常用的基因导入载体。pSET152图谱如下:
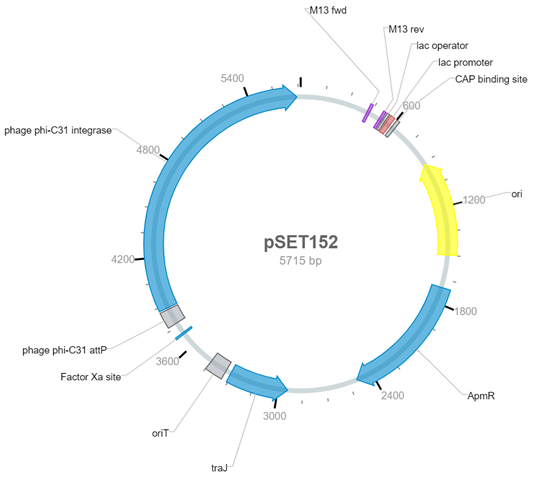
Souce: 纽普生物 2024-09-05