RNA 剪接(RNA splicing)是真核生物 RNA 转录后加工的必要环节,是导致转录组多样性和复杂性的关键步骤。真核细胞的基因序列中,包含了内含子(intron)与外显子(exon),两者交互穿插。其中内含子在基因转录成mRNA前体后会被RNA剪接体移除,剩下的外显子才是能够存在于成熟mRNA(之后再进一步翻译成蛋白质)的片段。
为了明确细胞核内含子剪接的分子机制,我们必须清楚剪接位点的特点。通过比较mRNA和相应结构基因的核苷酸序列,发现内含子的两端之间并没有很强的同源性或互补性,然而,剪接位点序列尽管非常短,但是仍有极端保守的共有序列(如图1)。在每一个共有位置处,每一个字母的高度代表特化碱基出现的百分率,这样,在紧邻的假定内含子接界处,可以发现高度保守的序列,其通用的内含子序列为: GU…AG
因为内含子两端剪接位点的识别是根据起始点(二核苷酸 GU)、终止点(二核苷酸 AG)来确定的,所以剪接位点的这种特征常常称为GU-AG规则(GU-AG rule)(当然,DNA 的编码链序列为GT-AG)。
由于两个剪接位点的序列不同,所以它们可以定义内含子的两个末端的方向。它们的命名沿着内含子从左向右的方向,左边的称为5'剪接位点(5'splice site),又称为左侧剪接位点(let site)或供体位点(donor site,SD);右边的称为3'剪接位点(3'splice site),又称为右侧剪接位点(right site)或受体位点(acceptor site,SA)。剪接位点的突变在体内和体外实验中都可以阻止剪接反应的进行,所以可进一步证明此共有序列参与了剪接事件。除了基于GU-AG规则的主要内含子,有机体还存在少数内含子特例,它们在外显子-内含子边界处拥有一组不同的共有序列,如图1的下部所示。这些内含子就是最初被描绘成基于AU-AC规则的次要内含子,这是因为在每一个内含子的两个末端中拥有保守的 AU-AC 二核苷酸,如图1的中部所示。
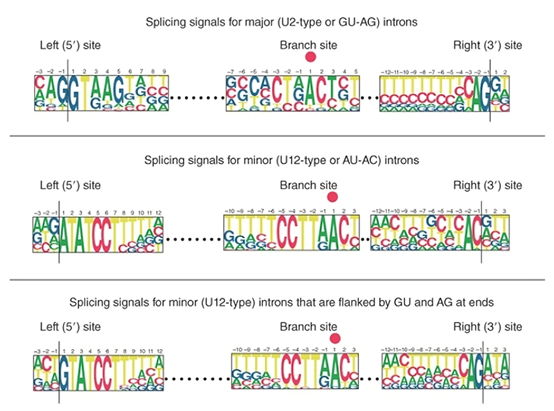
图1. 主要内含子(U2)和次要内含子(U12)的结构和序列特征
内含子的剪接基本过程(图2):外显子与内含子 5′ 端 GU 之间的磷酸二酯键发生裂解, snRNP(U1)含有与 5′剪接位点互补的序列,并与之结合;snRNP(U2)与分支位点结合,吸引 U1,游离的 5′ G 与分支位点的 A 之间的磷酸二酯键形成长链;第二个外显子与内含子的 3′ AG 之间的磷酸二酯键裂解;内含子被移除,两个外显子连接在一起。
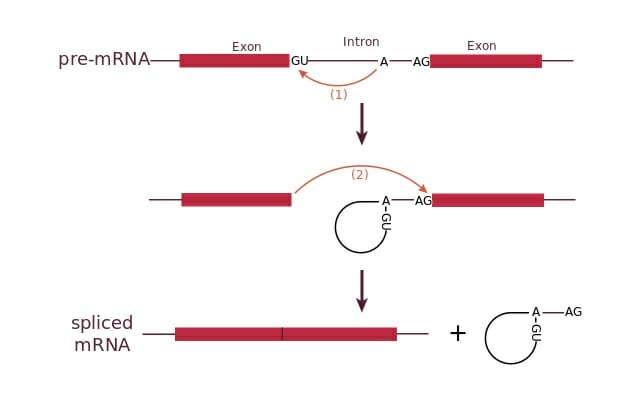
图2. 内含子的剪接过程
外显子捕获(Exon Trapping)
外显子捕获技术(Exon Trapping)是一种用于识别和捕获转录序列(即外显子)的方法,核心思想是利用外源DNA的RNA剪接能力,将目标外显子整合并捕获到已知的基因组片段中,从而有效识别潜在的基因。
具体而言,外显子捕获技术的过程可以简化为以下几个步骤:
-
克隆与载体构建:将待检测的基因组DNA片段克隆到一个经过修饰的载体中。这个载体通常携带有一个剪接供体位点(SD)和一个多克隆位点(MCS),目标DNA片段被插入到这个多克隆位点处。
-
转染与表达:将构建好的载体转染到哺乳动物细胞中,进行转录和剪接。插入的DNA片段如果含有功能性的外显子剪接受体位点(SA),则在转录过程中会发生剪接,去除内含子。
-
外显子捕获:转录出的RNA经过剪接并逆转录为cDNA,携带捕获的外显子。这些cDNA通过PCR扩增或其他方式回收,以分析外显子与已知供体位点之间的连接情况。
-
筛选与分析:通过DNA测序或杂交技术来验证外显子捕获事件。真正的外显子捕获事件表现为特定的外显子与供体位点正确连接,表明该外显子可能是某个基因的一部分。
这种技术的最大优势是能够有效从长段的基因组DNA中筛选出编码区域(外显子),尤其适用于尚未完全注释的基因组区域。
接下来我们以一篇论文[1]为案例讲解外显子捕获,和上面描述的略有点差异。
论文中,作者构建了一个pSPL3的质粒,图谱如下:
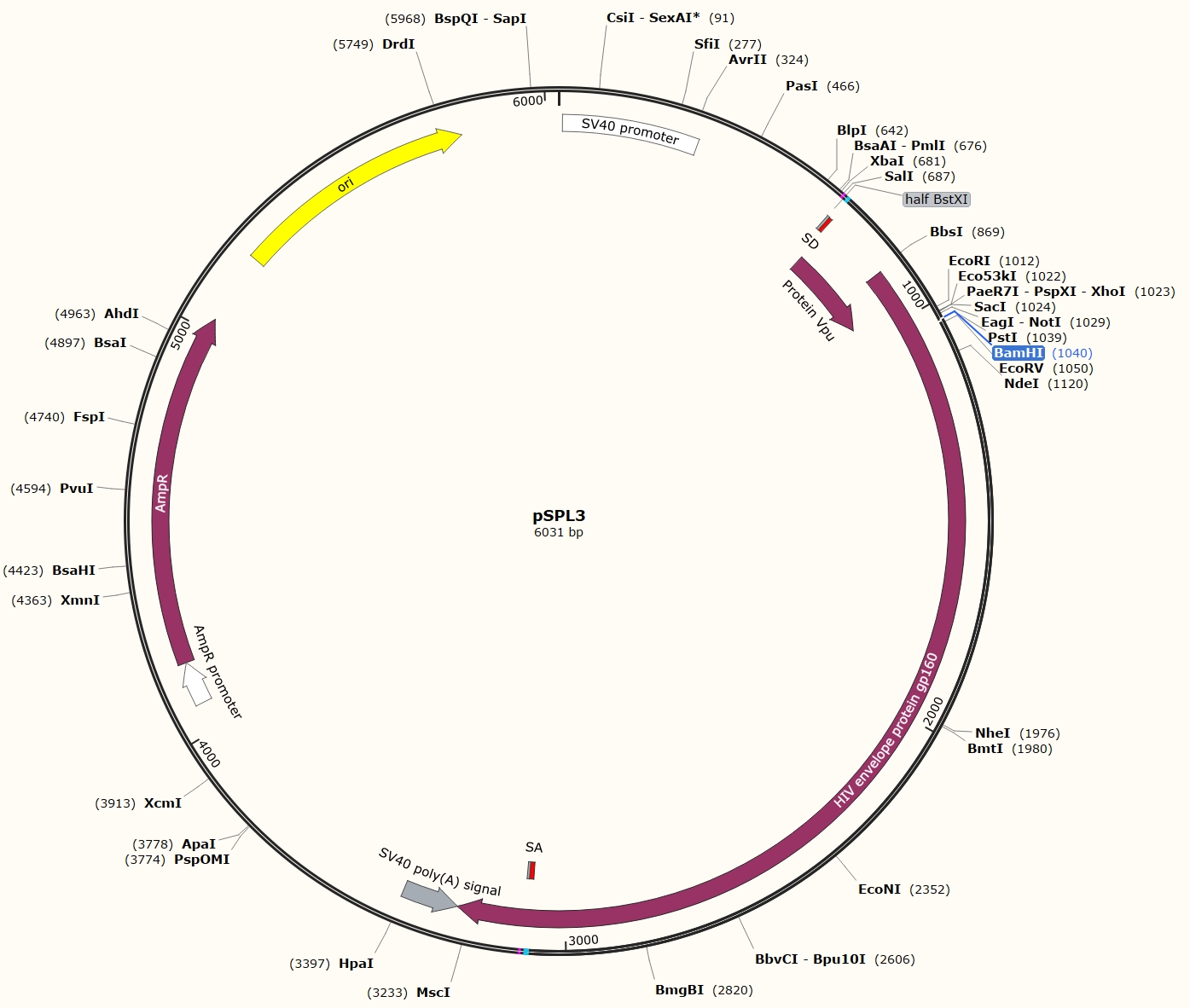
这个质粒包含以下几个关键元件:
SV启动子和polyA signal,实现目的片段在哺乳细胞中的转录和剪接。
SD和SA:内含子的SD和SA序列
两个half BstXI序列:分别为ccagca和acctgg,BstXI 限制性内切酶可识别 CCANNNNNNTGG 位点。另外,HIV gp160区域包含多个BstXI 限制性内切酶识别位点。
BamHI位点:位于SD和SA之间,与BglII内切酶为同尾酶(都产生相同的粘性末端:gatc)
实验过程:
1. 将目标基因组 DNA用 BamHI 和 BglII双酶切,pSPL3质粒用BamHI单酶切并去磷酸化(目的是为了防止载体自连接)。基因组DNA片段与pSPL3连接,连接产物转化到大肠杆菌中。将产生的菌落进行质粒 DNA 提取。提取的质粒 DNA 用于转染 COS7 细胞。
2. 转染 48-72 小时后,提取mRNA,并进行第一链 cDNA 合成。然后,使用特定引物进行PCR 扩增,将单链DNA转化为双链 DNA。为了消除仅含载体和假阳性的产物,加入 BstXI 进行消化,并进行第二轮 PCR 扩增。
3. 将PCR 产物用 SalI 和 BglII 酶切,然后克隆到经 SalI 和 BamHI 酶切的 pBluescriptIIKS+ 中。对克隆进行测序和序列分析。
当基因组片段中包含完整的外显子及侧翼内含子序列时,该外显子会被保留在成熟的polyA+ mRNA 中,通过 RNA-based PCR 扩增可检测到该外显子的存在,从而实现外显子的捕获。
参考文献
Church DM, Stotler CJ, Rutter JL, Murrell JR, Trofatter JA, Buckler AJ. Isolation of genes from complex sources of mammalian genomic DNA using exon amplification. Nat Genet. 1994 Jan;6(1):98-105. doi: 10.1038/ng0194-98. PMID: 8136842.
Souce: 纽普生物 2024-09-21